Dei siste hundre åra har forskarar funne ut stadig meir om korleis alle levande organismar er bygde opp, og ein har utvikla genteknologi for å undersøkje eller endre arvestoffet til ein organisme. Genteknologi som gjer det mogleg å studere enkeltgen og gjere endringar i arvestoffet, har vore i bruk sidan midten av 1970-talet. Det opna ei heilt ny verd av moglegheiter for å forstå og kontrollere biokjemiske prosessar.
I dag bruker ein genteknologi på forskjellig vis både på menneske, dyr, plantar og mikroorganismar. På denne temasida skal vi forklare nokre av basismetodane innan genteknologi som blir brukte i forsking og nokre av dei store oppdagingane det har ført til, men først må vi sjå på DNA-molekylet. Du kan lese meir om korleis denne teknologien har blitt utvikla på temasida Genteknologi som endrer gener: fra genspleising til genredigering.
Arvestoffet – DNA
Arvestoffet vårt, DNA-et, er bygd opp av fire grunnleggjande byggjesteinar: basane adenin (A), cytosin (C), guanin (G) og tymin (T). Desse basane er kopla saman på lange trådar, og det er rekkjefølgja av desse basane som bestemmer den genetiske koden. To trådar er tvinna om kvarandre slik at det blir danna basepar mellom basane på dei to trådane. Trådane dannar ein dobbeltspiral, også kalla ein DNA-dobbelheliks.
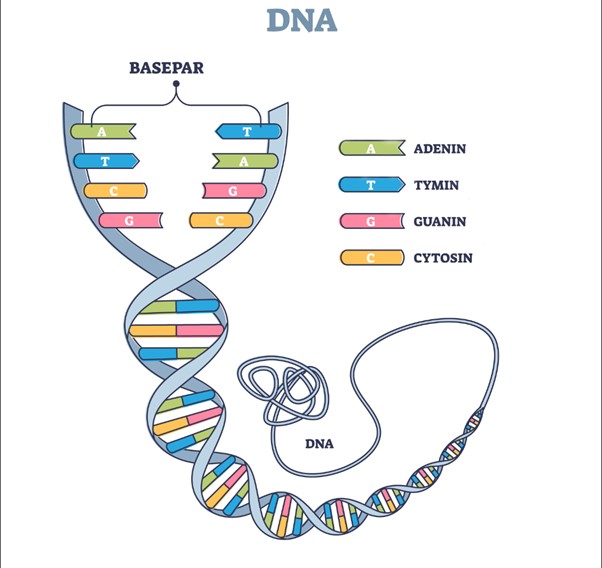
Alle organismar i verda er bygde opp av dei same byggjesteinane, frå bakteriar til plantar, fisk og menneske. Eit gen er eit stykke av eit DNA-molekyl der rekkjefølgja av basar kodar for eit protein (sjå temaside arv og genetikk). I menneske utgjer gena mindre enn to prosent av det totale DNA-et. Dei resterande 98 prosent kallar vi for ikkje-kodande DNA. Protein er bygde opp av lange rekkjer av aminosyrer, og det er proteina som utfører dei ulike jobbane i cellene. Tre og tre basar i DNA-et kodar for ei aminosyre i proteinet og totalt finst det 20 ulike aminosyrer. Då ein lærte seg å lese denne koden på slutten av 1960-talet, trudde ein at denne koden var universell, at dei same tre bokstavane kodar for den same aminosyra i alle organismar. Sjølv om det seinare har vist seg at det finst nokre unntak frå denne regelen, er koden godt bevart mellom ulike artar. Det gjer det mogleg å setje gen frå éin organisme inn i ein annan organisme og få genet til å lage proteinet det kodar for, i den organismen òg. I tillegg gjer denne likskapen mellom organismar det mogleg å studere for eksempel genfunksjonar i éin (modell-) organisme og generere ny kunnskap som gjeld generelt i andre organismar.
Isolering av DNA
I dag er det mogleg å isolere og kartleggje rekkjefølgja til alle basane i heile arvestoffet (genomet) til ein organisme. Dette har mange viktige bruksområde innan forsking. Analysar av arvestoffet kan mellom anna brukast til å finne genetisk årsak til sjukdom hos menneske, eller for å forstå meir om gena sin funksjon hos ulike organismar. Ein nyttar då ein biologisk prøve, og isolerer DNA frå denne biologiske prøven. Biologiske prøvar kan vere celler frå ulike typar vev. Hos menneske bruker ein ofte blod eller celler frå innsida av munnen.
For å isolere DNA-et i den biologiske prøven må ein først øydeleggje cellene, slik at DNA-et blir tilgjengeleg. Dette gjer ein ved å blande cellene med såpe. Såpa øydelegg cellemembranane, som består av feitt. Ein må så tilsetje eit enzym som øydelegg alle proteina i cellene. Dette for å unngå at nuklease-enzyma i cellene, protein som kuttar DNA, ikkje øydelegg DNA-et når det kjem ut av cellekjernen. Til slutt må ein skilje DNA-et frå alt det andre i cellene, slik at ein sit att med ei løysning som berre inneheld DNA. Dette gjer ein ved å tilsetje alkohol (for eksempel etanol). DNA-et samlar seg då i sjiktet mellom alkoholen og resten av løysninga, og flokar av DNA-trådar blir synlege for auget som kvite klumpar. Dette DNA-et kan ein så løyse i ei eigna væske (ein buffer) som gjer det stabilt.
Kopiere DNA med PCR
Det kan vere som å leite etter ei nål i ein høystakk å skulle få tak i det DNA-området ein skal arbeide med, då eit gen berre er ein ørliten liten del av eit stort kromosom. Etter at vi fekk PCR-metoden i 1986, har dette arbeidet blitt mykje lettare. Ved hjelp av PCR (Polymerase Chain Reaction – polymerase kjedereaksjon) får ein i løpet av kort tid ekstremt mange kopiar av akkurat det DNA-området ein er ute etter. For å kunne bruke PCR-metoden må ein kjenne til baserekkjefølgja til delar av DNA-området ein skal kopiere (figur 2).
Kopiering av DNA med PCR blir mykje brukt i forsking når ein for eksempel skal få tak i ein kopi av eit gen ein forskar på , eller når ein skal analysere andre delar av DNA-et . Dersom ein for eksempel ønskjer å lage ein DNA-profil for å identifisere ein person, bruker ein PCR for å få mange kopiar av dei DNA-områda som skal analyserast (sjå temasida om gentesting og genetiske undersøkingar).
PCR blir også nytta for å analysere for smittsame sjukdommar, som kan stadfeste om ein er smitta med for eksempel koronavirus eller om ein ny virusvariant har utvikla seg. Når ein skal teste for koronavirus, nyttar ein PCR-metoden til å lage mange kopiar av koronaviruset sitt arvemateriale, som er heilt unikt.
Rekombinant DNA teknologi – klippe og lime DNA
PCR-metoden har revolusjonert korleis vi kan kopiere opp bitar av DNA, men før PCR-metoden blei introdusert i 1986, nytta forskarar andre metodar for å kunne studere små bitar DNA for seg. Då sat dei på laboratoriet og klipte opp dei lange DNA-trådane i små bitar og sette desse inn i organismar som laga mange kopiar av dei utvalde DNA-bitane, det som heiter rekombinant DNA-teknologi. Rekombinant DNA-teknologi er nyttig i dag òg for å forstå funksjonen til ulike gen eller protein.
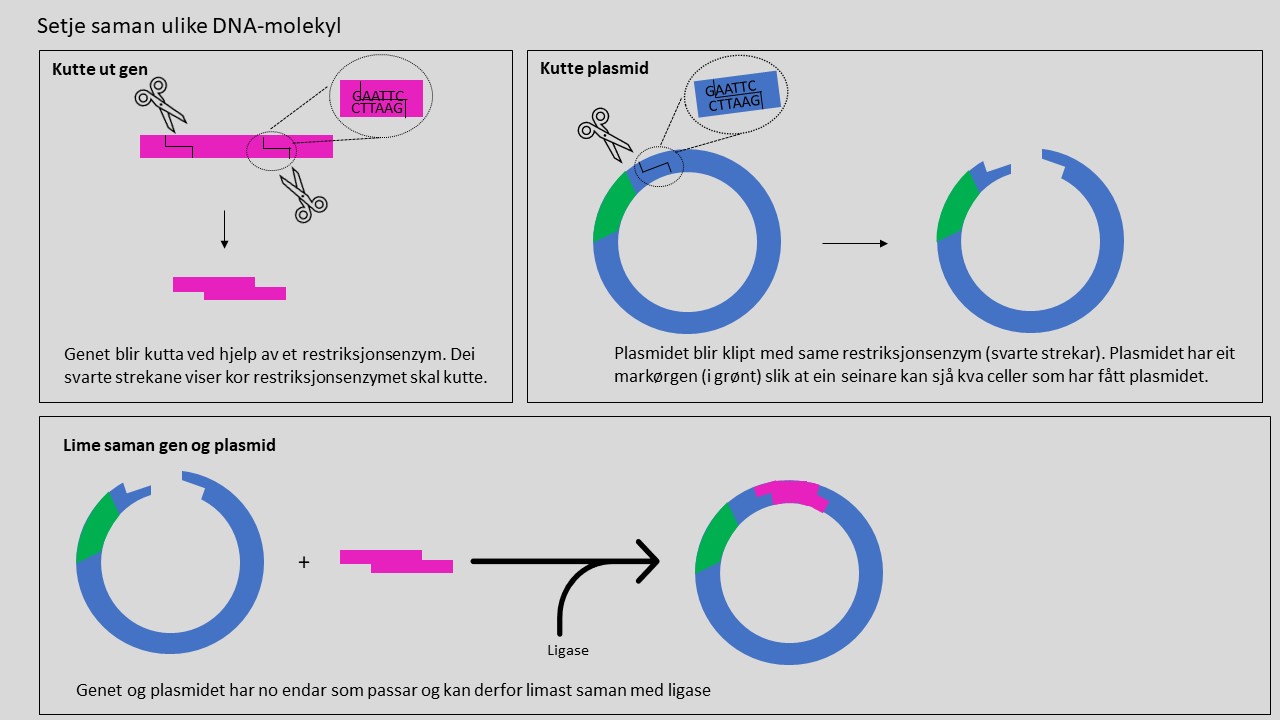
Når ein skal setje saman ulike DNA-bitar frå ulike organismar, må ein klippe og lime DNA-et. Det kan for eksempel vere når ein bruker bakteriar til å lage insulin til menneske. Då set ein eit gen frå menneske som kodar for insulin inn i eit plasmid (eit sirkulært DNA-molekyl som bakteriar kan ha) som etterpå blir sett inn i ein bakterie. (Sjå temaside om industriell bioteknologi).
For å lage rekombinant DNA må ein gjennom fleire trinn. Først må ein behandle DNA-et for å lage DNA-bitar som passar saman. Det er vanleg å klippe/kutte DNA-et slik at den eine DNA-tråden blir lengre enn den andre i endane (figur 3). Dette gjer ein ved å blande DNA-et med «molekylære sakser» som blir kalla restriksjonsenzym. Dette er ulike protein som kjenner igjen spesifikke DNA-sekvensar og klipper DNA-molekylet ved desse sekvensane. Ei rekkje ulike restriksjonsenzym finst naturleg i bakteriar, der dei vernar bakteriane mot ukjent DNA frå for eksempel virus.
Dersom ein ønskjer å kople saman ulike DNA-bitar, bruker ein eit «molekylært lim» som blir kalla ligase. Dette er eit enzym som kan binde saman DNA-bitar som passar saman. Ligasar finst naturleg, då dei har som «jobb» å reparere DNA-skadar i organismar.
Gelektroforese
Når ein kuttar DNA, arbeider ein med svært små mengder DNA og enzym, og ein kan ikkje sjå kva som skjer med DNA-et direkte. Ein må derfor analysere det oppkutta DNA-et. Det kan ein gjere ved å køyre gelelektroforese på DNA-prøvane, ein metode som skil DNA-bitar frå kvarandre etter størrelse. Ein kan bruke ein gel (ei geléliknande plate) som er laga av agarose eller polyakrylamid. Denne gelen er full av porer som gjer at små molekyl lett kan vandre gjennom, mens store molekyl blir bremsa. I gelen plasserer ein DNA-prøvar i brønnar (figur 4). Prøvane er blanda med eit fargestoff som bind seg til DNA og som gjer at ein kan sjå kvar i gelen DNA-et er (sjå bilde). Gelen ligg i eit kar i ei væske som inneheld salt, slik at han leier straum. I karet har ein elektrodar, og gelen ligg slik at brønnane ligg ved den negative polen. Sidan DNA-et er negativt lada, vil DNA-bitane vandre mot den positive polen når ein set på straum. Etter ei tid vil dei små DNA-bitane ha vandra langt, mens dei store bitane har vandra kort.

DNA-sekvensering – å lese DNA
Å sekvensere DNA inneber å kartleggje rekkjefølgja på basane i DNA-molekylet, eller delar av DNA-molekylet, og dermed få tilgang til ein organisme sin genetiske kode. I dag gjer sekvenseringsteknologi det relativt lett å bestemme alt frå sekvensen til enkeltgen i ulike individ (genvariantar) til heile den genetiske koden (genomet) til ein organisme. Derfor er sekvensering særs mykje brukt i forsking. Informasjon om rekkjefølgja av basane kan blant anna brukast til å forstå korleis gena våre er bygde opp og fungerer, kvifor menneske er forskjellige seg imellom til korleis ulike artar har utvikla seg i evolusjonen (inklusiv artar som er utdøydde). Innan medisin er informasjonen frå DNA-sekvensering blant anna viktig for å forstå og diagnostisere ei rekkje genetiske sjukdommar, og for nokre genetiske sjukdommar er sekvensering viktig for å gi rett behandling (sjå temaside om genterapi).
Det var på 70-talet at den britiske biokjemikaren Frederick Sanger oppfann ein metode for å lese DNA-sekvensen. Metoden blir gjerne kalla Sanger-sekvensering, dideoksysekvensering eller førstegenerasjonsekvensering (sjå faktaboks). I 1977 brukte Sanger og kollegaer den nye metoden for å lese heile genomet til ein organisme for første gong, viruset Phi-X174. Genomet til Phi-X174-viruset består av rundt 5300 DNA-basar, som var ei enorm bragd å sekvensere den gongen. Til samanlikning består det menneskelege genomet av omkring 6,4 milliardar basepar, noko som lenge var heilt urealistisk å sekvensere.
I 1990 vart humangenomprosjektet (HGP) lansert. Dette var ei stor internasjonal satsing og involverte fleire tusen forskarar frå meir enn 80 land. Prosjektet hadde som mål å kartleggje heile menneskets DNA-sekvens, altså rekkjefølgja på alle basane i DNA-molekylet. Den første versjonen av mennesket sitt genom blei publisert i 2001 og blei offisielt ferdigstilt i 2003. Då hadde tusenvis av forskarar i heile verda brukt over 10 år og 300 millionar dollar på å kartleggje rundt 90 prosent av det menneskelege genomet. I dei seinaste åra har ein brukt mykje ressursar på gjere kartlegginga fullstendig. I 2022, omkring 30 år etter lanseringa av humangenomprosjektet, tok forskarar eit stort steg vidare ved å sekvensere det gjenståande DNA-et. Dette var mogleg blant anna takk vere ein ny sekvenseringsteknologi; «long read sequencing» (sjå faktaboks). I takt med at teknologien er blitt betre, har òg kostnaden for å sekvensere DNA falle betrakteleg.
Genomet til dei fleste organismar er for langt til å kunne lesast som ein enkelt tråd. For å kunne lese rekkjefølgja på basepara, må DNA-et framleis først kuttast i små bitar. Kvar bit blir lesen av ein sekvenseringsmaskin, og deretter må dei ulike bitane setjast saman. Førestill deg ein haug med fleire tusen bøker, der alle sidene blir klipte opp i små bitar. Deretter skal alle bitane lesast og setjast saman igjen, slik at alle ord kjem i riktig rekkjefølgje, som eit gigantisk puslespel. Jo fleire sider det er i boka, desto vanskelegare blir det å setje bitane saman. I tillegg til sjølve sekvenseringteknologien har datateknologi vore heilt sentralt i det at vi i dag kan setje saman og samanlikne heile genom til ulike organismar. Slik bioinformatikk, som er eit felt der ein bruker spesialprogramvarer og superdatamaskinar til å behandle biologiske data, er svært viktig for å kunne tilverke og analysere komplekse sekvensdata.
Genomikk
Genomikk er eit forskingsområde der heile genomsekvensar blir analyserte, og ein kan kartleggje korleis ulike gen interagerer med kvarandre og omgivnadene. Dette kan for eksempel vere å samanlikne genomet til ulike individ og ulike organismar, både eksisterande og utdøydde.
Sjølv om det i dag er mogleg å sekvensere heile genomet til alle individ av kvar ein organisme, er det framleis mykje vi ikkje forstår om korleis alle gena våre fungerer saman.
Såkalla funksjonell genomikk er forsking på funksjonane til gena i ein organisme. Kjenner ein funksjonane til eit gen frå ein organisme, kan sekvensering og bioinformatikk fortelje noko om funksjonen til liknande gen i andre organismar. Ein metode som også i dag blir brukt mykje i funksjonell genomikkforsking, er genredigering med Crispr-metoden (sjå temaside om genredigering og Crispr). Det er ein metode der ein kan endre sekvensen i eit gen, for eksempel i ein cellekultur eller ein modellorganisme. Ein kan då endre eller «slå ut» eit gen og studere funksjonen til genet ved å sjå kva som skjer. Fleire av dei biologiske prosessane er universelle: for eksempel bruker ein i dag gjennomsiktige akvariefisk (sebrafisk) til å forske på ulike gen sin funksjon i menneskets nervesystem og for å undersøkje om ei endring av gensekvensen i eit gen kan gi sjukdom.
Genteknologi og jakta på sjukdomsgen
I dag kan heile det menneskelege genomet bli sekvensert på nokre få døgn og enkeltgen på nokre timar, men det har ikkje alltid vore slik. Før nyare sekvenseringsteknologi blei utvikla, var det mykje meir tid- og ressurskrevjande å identifisere sjukdomsgen.
Eit eksempel er historia om «brystkreftgena» BRCA1 og BRCA2 som blei oppdaga før dagens sekvenseringsteknologi. I dag veit vi at arveleg bryst- og eggstokkreft oftast kjem av feil i desse gena. Jakta på brystkreftgenet starta på 40-talet, då det gjekk opp for forskarar at brystkreft var ein sjukdom som nokre gonger gjekk i familiar. I takt med at forskarar fekk betre forståing av kva DNA er og korleis gen fungerer, blei det stadig tydelegare for dei at i nokre familiar så var brystkreft knytt til defekte gen. Men det tok nokre tiår før ein klarte å identifisere kva gen som var knytte til sjukdommen. For å identifisere kor i arvematerialet genet var, brukte forskarane genetiske koplingsundersøkingar. Dette er undersøkingar der ein studerer korleis eit gen blir nedarva saman med sjukdommen innanfor ein familie. I 1994, etter fleire år med intens forsking, klarte dei endeleg å isolere BRCA1-genet og vise at nokre mutasjonar i dette genet var hyppig i familiar med arveleg bryst- og eggstokkreft. Sjølv om det enno er utfordrande å identifisere nye sjukdomsgen, vil forskarar i dag ha ei heilt anna tilnærming som er langt meir effektiv enn tidlegare. I dag kan ein for eksempel samle DNA frå fleire pasientar med same symptom og som ein mistenkjer har same sjukdom. Deretter vil DNA-prøvane bli analysertee for å identifisere kvar ein eventuell mutasjon kan vere. Ein kan for eksempel sekvensere heile genomet (heilgenomsekvensering) eller alle gena (heileksomsekvensering) i ein prøve for å sjå etter mutasjonar.
Paleogenomikk og jakta på utdøydde genom
Paleogenomikk er eit forskingsfelt som handlar om genetiske analysar av fossile og prehistoriske leivningar. I takt med at dei genteknologiske metodane har blitt betre, har paleogenomikk-feltet òg utvikla seg enormt.
Mens nokre på 90-talet jobba med å kartleggje det humane genomet, var andre på jakt etter genomet til utdøydde menneskeartar. Ein av desse er den svenske genforskaren Svante Pääbo. I oktober 2022 fekk han Nobelprisen i medisin for sitt arbeid rundt menneskeleg evolusjon og for sin innsats for å genomsekvensere våre fjerne slektningar; neandertalarane og denisovanarane. Han og kollegaene klarte å sekvensere DNA frå eit 40 000 år gammalt bein og fann fram til ein ukjend menneskeart, denisovanaren. Forskinga er ei viktig puslebrikke i forståinga av korleis menneskearten har utvikla seg over tid. Det er med utgangspunkt i denne forskinga at vi no veit at menneske med europeisk eller asiatisk avstamming har rundt éin til fire prosent DNA som stammar frå neandertalarane og at forfedrane våre derfor sannsynlegvis har fått barn med neandertalarane for fleire tusen år sidan. Analysar av eldgamle DNA-prøvar kan også brukast for å forstå korleis menneske har flytta på seg over tid og har bidratt til å løyse store gåter om menneskets historie. For eksempel prøver forskarar å estimere når dei første menneska innvandra til Amerika ved hjelp av DNA-analysar av fleire tusen år gamle bein og restar av avføring. Eit anna eksempel er ein nyare studie der forskarar har kasta lys på korleis menneske frå Danmark og andre nordeuropeiske land fann vegen til England lenge før vikingtida.

Ny kunnskap om bakteriar og virus
Genteknologi og sekvensering er også viktig for å lære meir om bakteriar og virus, som for eksempel kvifor dei har visse eigenskapar og korleis desse organismane utviklar seg.
Ved å sekvensere koronavirusets genom, har forskarar for eksempel kunna følgje med på kva nye genetiske endringar som oppstår i koronavirusets arvemateriale over tid, noko som har vore viktig for å overvake når nye virusvariantar oppstår. Slik informasjon kan for eksempel brukast til å optimalisere metodane for diagnostikk og påvise viruset hos menneske.
Genomsekvensering er også eit viktig verktøy for å forstå bakteriebiologi, for eksempel korleis dei utviklar resistens mot antibiotika. Antibiotikaresistens er evna bakteriar har til å motstå effekten av antibiotika og er ein trussel for framtidas medisin. Sekvensering av bakteriane sitt genom er ein sentral reiskap i kampen mot antibiotikaresistens: det bidrar blant anna til forståinga av korleis bakteriane blir resistente mot antibiotika, og kunnskapen blir nytta til å overvake spreiing av antibiotikarestistente bakteriar og til å utvikle nye typar antibiotika.
På veg mot fullsekvensering av alle organismar?
I løpet av dei siste 25 åra har genomikk-studiar vore eit viktig verktøy i forsking på plantar og dyr. Det første dyret som blei sekvensert, var rundormen (Caenorhabditis elegans) i 1998. Etter kvart som genom av andre dyr er blitt fullsekvensert og analysert, har forskarar fått innblikk i dei mange genetiske variasjonane som skil dei ulike dyra og kva betydning desse har for biodiversiteten på jorda. No har forskarar ambisjonar om å genomsekvensere alle kjende organismar på jorda.
Slike heilgenomanalysar kan gi verdifull informasjon om evolusjonen: korleis mikrobar, plantar og dyr har utvikla og endra seg over tid og korleis evolusjonen går for seg akkurat no. I tillegg er slik informasjon med på å byggje eit viktig kunnskapsgrunnlag som for eksempel kan bidra til å utvikle nye medisinar, optimalisere landbruket og finne løysingar på korleis vi bevarer og beskyttar trua artar.
Utvikling av DNA-sekvenseringsteknologiar
I seinare tid har det skjedd ei rekkje nyvinningar innan sekvenseringsteknologi. I dag skil ein i hovudsak mellom tre typar sekvensering.
1970-talet: Sanger-sekvensering (førstegenerasjonssekvensering): Dette er sett på som den første DNA-sekvenseringsmetoden og blei utvikla på 70-talet av forskaren Frederick Sanger. Metoden blei sett på som banebrytande og resulterte i at Sanger fekk Nobelprisen i kjemi i 1980. Sanger-sekvensering blir framleis i dag brukt til å analysere kortare sekvensar, som for eksempel DNA som er kopiert med PCR, men er langt mindre effektiv og dyrare samanlikna med nyare sekvenseringsmetodar. Derfor blir ikkje sanger-sekvensering brukt til større prosjekt, som for eksempel heilgenomsekvenseringar.
Tidleg 2000-talet: Nestegenerasjonssekvensering (andregenerasjonssekvensering): Ei av dei store nyvinningane var utviklinga av nestegenerasjonssekvensering (NGS), også kalla massiv parallell sekvensering. Ved denne metoden kan ein lese millionar av DNA-basepar samtidig. Dette gjorde at sekvensering blei mykje raskare og billigare enn før.
2008–: Long-read sequencing (tredjegenerasjonssekvensering): Long-read sequencing er ein nyare sekvenseringsmetode. Ved denne metoden kan ein lese mykje lengre DNA-sekvensar på ein gong (opptil 100 000 basepar). Fordelen ved denne metoden er at det er lettare å setje saman bitane etter sekvensering. I tillegg gjer metoden det lettare å analysere repetitive regionar (regionar der same rekkjefølgje på basane opptrer fleire gonger etter kvarandre).
Å lage ein genmodifisert organisme
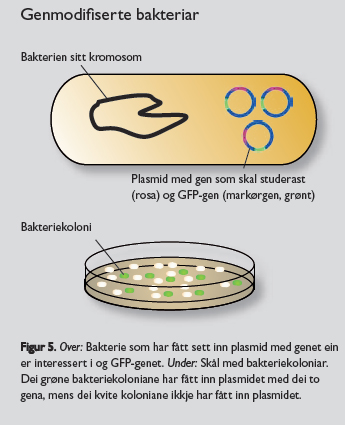
Genmodifiserte organismar er plantar, dyr eller mikroorganismar som har fått arvestoffet endra ved hjelp av genteknologi. Genmodifiserte organismar blir mykje brukte i forsking. For eksempel kan ein genmodifisere ein bakterie for å få den til å produsere eit bestemt protein (for eksempel ved rekombinant DNA-teknologi). Dette kan vere nyttig for å isolere proteinet og studere og analysere proteinstrukturen. Dette kan òg vere nyttig for å produsere store mengder av protein, som for eksempel insulin (sjå temaside om industriell bioteknologi). Dersom ein ønskjer å få ein bakterie til å produsere eit bestemt protein, må ein lage eit plasmid som inneheld genet som kodar for det aktuelle proteinet (figur 3). Plasmidet med genet blandar ein så med bakteriar under slike forhold at plasmidet kjem inn i bakteriane (figur 5). For at ein etterpå skal kunne sjekke at ein verkeleg har fått genet inn i bakterien, er det vanleg å ha eit såkalla markørgen i plasmidet. Ved hjelp av dette markørgenet kan ein lett finne dei bakteriane som har tatt opp plasmidet med dei nye gena.
Markørgenet kan for eksempel vere eit gen for antibiotikaresistens. Då dyrkar ein bakteriane med antibiotika til stades, slik at berre dei bakteriane som har fått antibiotikaresistensgenet, klarer å vekse opp. Dei bakteriane som har fått markørgenet, har òg fått genet for proteinet ein er interessert i. Markørgenet kan òg vere eit gen som gir bakteriane farge. Det er for eksempel mogleg å bruke eit gen frå manetar, som kodar for eit «sjølvlysande» protein (grønt fluorescerande protein – GFP). Bakteriar som får dette manetgenet, blir grøne når det blir produsert protein frå genet (figur 5).
Når ein har valt ut bakteriar som har dei gena ein ønskjer, og som ein veit produserer det ein vil, dyrkar ein desse bakteriane i store mengder. Deretter reinsar ein produktet ut frå «bakteriesuppa» ved hjelp av biokjemiske metodar.
Spørsmål til diskusjon
Bruk av genteknologi der ein endrar på gen i organismar for eksempel for å studere sjukdomar, er svært nyttig i forsking, men reiser òg etiske problemstillingar:
- Er det rett å endre på gena til dyr for å forske fram ny kunnskap om menneske?
- Er det det forskjell på kva som er etisk forsvarleg å gjere i forsking på sjukdommar hos menneske og sjukdommar hos dyr?
- Er det forskjell på å bruke sebrafisk og mus som forsøksdyr i forsking?
Metodane for å lese og analysere DNA-sekvensen blir stadig betre. Utviklinga gir oss ei rekkje nye moglegheiter, men reiser òg fleire etiske spørsmål, for eksempel:
- Kva for problemstillingar synest du er knytt til det å dele genetisk informasjon om seg sjølv i eit forskingsprosjekt? Er det for eksempel relevant at alle delvis har mykje felles genetikk med nær slekt (foreldre/søsken/eigne barn)?
Innhaldet på denne sida vart sist oppdatert i september 2024.
Send oss ein e-post om du har spørsmål eller kommentarar til innhaldet.